Dem Chemiker Ivan Huc von der Ludwig-Maximilians-Universität München (LMU) ist es gelungen, Eigenschaften der Oberfläche einer DNA-Doppelhelix so nachzuahmen, dass Proteine mit dem Imitat interagieren.
Meist gilt das Orginal als die bessere Version. Der Forscher Ivan Huc konnte dies nun wiederlegen. Er hat mit seinen Foldameren künstliche Moleküle geschaffen, an die Proteine sogar besser binden als an die Ursprungsvariante.
In der Studie blockierten die künstlichen Foldamere verschiedene Enzyme von Viren, darunter die HIV Integrase. Mit diesem Protein schleust der HI-Virus sein Genom in die Wirtszelle ein. Die Forschungsergebnisse von Ivan Huc und seinem Team könnten ganz neue therapeutische Ansatzpunkte eröffnen.
Der Wissenschaftler designt mit seiner Arbeitsgruppe „Biomimetic Supramolecular Chemistry“ an der LMU künstliche Moleküle, die sich mithilfe einer Art Origami-Technik nach dem Abbild ihrer natürlichen Vorbilder formen lassen. In der aktuellen Ausgabe dem Fachmagazin Nature Chemistry veröffentlichte Huc nun seine Forschungsergebnisse.
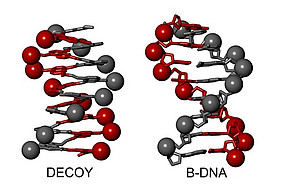
„Die Form bestimmt die Funktion“, erklärt Ivan Huc seinen Ansatz. In der nun neu erschienenen Studie hat der Chemiker als Basis ein künstliches Molekül entwickelt, das sich schraubenförmig falten und nach einer Art Baukasten-Prinzip vielfach modellieren lässt. So konnte Ivan Huc Oberflächeneigenschaften der natürlichen DNA-Doppelhelix imitieren. Dabei ist das Imitat so gut geworden, dass zwei Enzyme, darunter die HIV Integrase, auf die falsche DNA hereinfallen und dadurch blockiert werden können.
Das Imitat ist besser geworden als die echte DNA selbst und hat die Natur damit übertroffen: Die Bindung der HIV Integrase an das Foldamer war stärker als an die DNA selbst. „Obwohl das Design auf die Ähnlichkeit zur DNA abzielt, verdankt das Foldamer seine wertvollsten Eigenschaften gerade seinen Unterschieden zur DNA“, betont Huc.
Das Baukastenprinzip, nach dem sich die künstlichen DNA-Sequenzen nach Belieben gestalten lassen, eröffnet viele Variationsmöglichkeiten. In der vorliegenden Studie testete Ivan Huc die Funktion am Beispiel von Enzymen, die an eine beliebige Stelle der DNA binden. Es wäre jedoch auch denkbar, künstliche DNA-Abschnitte zu entwickeln, um Enzyme zu blockieren, die nur an bestimmte DNA-Sequenzen binden.
Weitere Informationen:
Pressemitteilung_DNA gekonnt imitiert, Virus ausgetrickst
zur Forschung von Ivan Huc: Ribosome tolerates artificial foldamers